Synthetische Polymere sind eine langlebige Alternative zur DNA
Eine Studie von Kyoung Taek Kim und seinem Team zeigt, wie synthetische Polymere zur langfristigen Datenspeicherung genutzt werden können. Durch innovative Fragmentierungs- und Sequenzierungsmethoden lassen sich große Polymerketten effizient auslesen und gezielt nach spezifischen Informationen durchsuchen. Diese Methode könnte künftig DNA (Desoxyribonukleinsäure) als Speichermedium in bestimmten Bereichen ergänzen.
Datenarchivierung mit Molekülen. Grafik: Wiley-VCH
Täglich entstehen große Datenmengen, sei es durch geschäftliche Transaktionen, Prozessüberwachung, Qualitätssicherung oder zur Rückverfolgbarkeit von Produktions-Chargen. Ihre Langzeitarchivierung erfordert erhebliche Speicherressourcen und Energie. Für die dauerhafte Speicherung umfangreicher Daten, die nur selten abgerufen werden müssen, bieten Makromoleküle wie DNA und synthetische Polymere eine sehr gute Alternative zu herkömmlichen Speichermedien, da sie gespeicherte Informationen mit deutlich geringerem Platz- und Energiebedarf aufrechterhalten.
Überraschung an der Korngrenze
Synthetische Polymere punkten im Vergleich zu DNA
Synthetische Polymere haben gegenüber DNA einige Vorteile, darunter eine einfachere Synthese, höhere Speicherdichte und bessere Stabilität unter anspruchsvollen Bedingungen. Ein Nachteil ist jedoch die begrenzte Speicherkapazität pro Polymerkette. Da die in Polymeren gespeicherten Informationen durch Massenspektrometrie (MS) oder Tandem-Massen-Sequenzierung (MS2) ausgelesen werden, müssen die Moleküle relativ klein bleiben, da die gesamte Kette schrittweise entschlüsselt wird. Dies ist vergleichbar mit einem Buch, das man vollständig durchlesen müsste, um eine spezifische Information zu finden. Bei DNA hingegen können lange Ketten in kürzere Fragmente aufgeteilt und anschließend rekonstruiert werden.
So leben Zink-Batterien deutlich länger
Lesbarkeit sehr langer synthetischer Polymerketten wurde verbessert
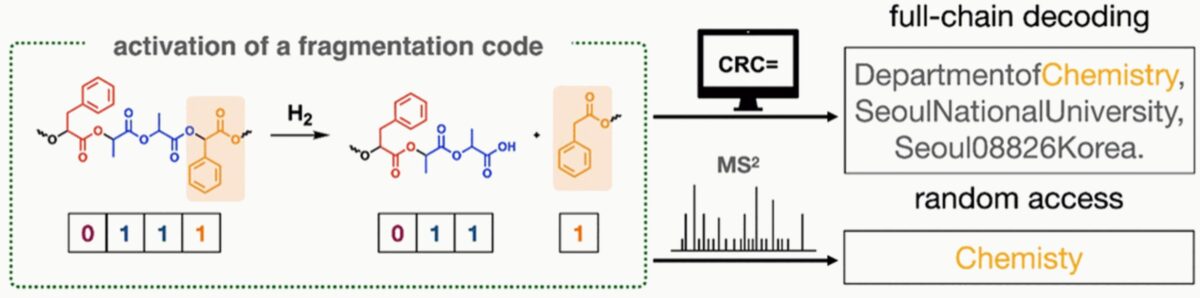
Ein Forscherteam unter der Leitung von Kyoung Taek Kim an der Seoul National University hat einen innovativen Ansatz entwickelt, um die Lesbarkeit sehr langer synthetischer Polymerketten zu verbessern, selbst wenn deren Molekulargewichte die analytischen Grenzen der MS- und MS2-Methoden überschreiten. Die Forscher kodierten beispielhaft die Universitätsadresse in ASCII und nutzten eine Prüfziffer (CRC-Code), um die Datenintegrität zu gewährleisten. Diese 512-Bit-Information speicherten sie in einer Polymerkette, die aus zwei Monomeren bestand: Milchsäure repräsentierte den Wert 1 und Phenyl-Milchsäure den Wert 0. Fragmentierungspunkte, die Mandelsäure enthalten, wurden strategisch in die Kette eingefügt, sodass sie sich bei Aktivierung in 18 Fragmente unterschiedlicher Länge aufspalten ließ, die einzeln per MS2 entschlüsselt werden konnten.
Eine speziell entwickelte Software identifiziert zunächst die Fragmente anhand ihrer Masse und Endgruppen. Bei der MS2-Methode werden die Molekülionen weiter fragmentiert und erneut analysiert, was eine Sequenzierung der Fragmente ermöglicht. Durch die Anwendung des CRC-Codes kann die Software die gesamte Sequenz präzise rekonstruieren, wodurch die bisherigen Längenbeschränkungen für Polymerketten überwunden werden.
Neue Berechnungsmethode revolutioniert die Simulation neuer Materialien
Relevante Fragmente werden präzise angesteuert
Darüber hinaus gelang es dem Team, spezifische Informationen in der Polymerkette zu lokalisieren und gezielt auszulesen, ohne die gesamte Kette sequenzieren zu müssen. So konnten sie beispielsweise das Wort „Chemistry“ in der kodierten Universitätsadresse selektiv extrahieren. Da die einzelnen Adressbestandteile durch Kommas getrennt und in einer festgelegten Reihenfolge angeordnet waren (Abteilung, Institution, Stadt, Postleitzahl, Land), war es möglich, die relevanten Fragmente präzise anzusteuern und nur diese zu sequenzieren.